急性心肌梗死(acute myocardial infarction,AMI)是由冠狀動脈病變引起的臨床病癥,可危及生命,是心臟性猝死的主要原因之一[1]。主要發病原因為冠狀動脈粥樣斑塊破裂,形成血塊阻塞動脈,導致心肌缺血壞死[2]。AMI可分為ST段抬高型心肌梗死和非ST段抬高型心肌梗死[3]。通常,心肌肌鈣蛋白T和心肌肌鈣蛋白I被作為診斷AMI的生物標志物。雖然心肌肌鈣蛋白的增加通常提示心肌壞死,但這也可能是由心力衰竭、心肌炎和胸部創傷等其他疾病引起[4]。所以,我們需要尋找新的診斷AMI的生物標志物,特別是在AMI早期具有高敏感性和特異性的分子,以縮短診斷期并改善AMI患者的預后。
鐵死亡(ferroptosis)是一種不同于細胞凋亡、細胞壞死和自噬的一種細胞死亡方式。主要與細胞內鐵的含量有關,表現為鐵含量過度升高并導致細胞內氧化還原反應失衡、脂質過氧化物的積累和活性氧的產生[5]。隨著冠狀動脈閉塞后血運重建引起再灌注損傷的發展,心肌細胞發生鐵死亡并釋放炎癥介質以加重心臟損傷[6]。有研究[7]發現阿霉素可誘導心臟中的血紅蛋白降解和游離鐵釋放,使心肌細胞發生鐵死亡,從而引發心力衰竭。尋找鐵死亡的新分子靶點正逐漸成為心血管研究領域的焦點。
1 資料與方法
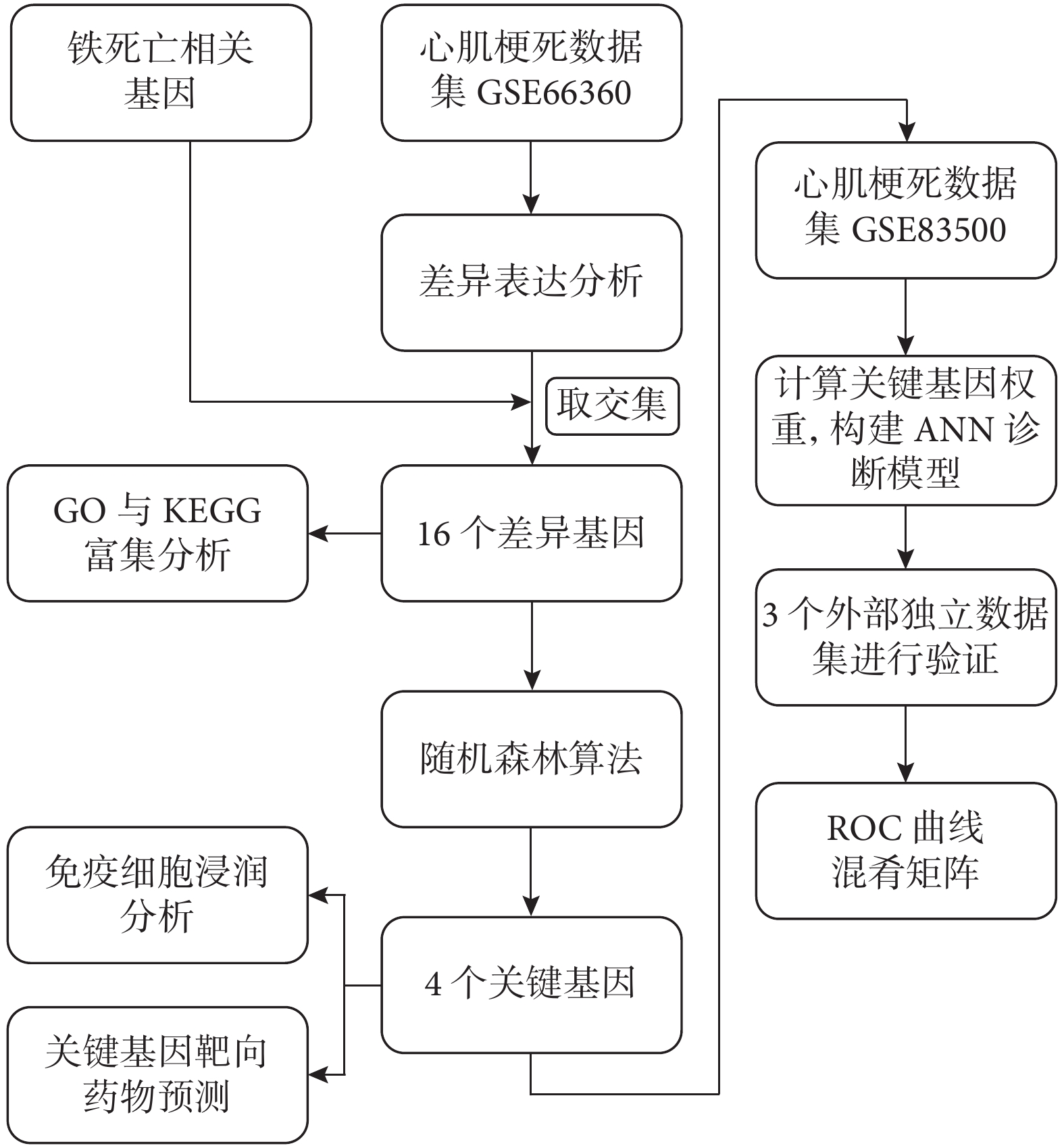
本研究從公共數據庫基因表達綜合數據庫(Gene Expression Omnibus,GEO)下載心肌梗死高通量測序數據,通過隨機森林算法篩選4個具有分類性能的心肌梗死鐵死亡特征基因。通過這4個基因構建人工神經網絡診斷模型。采用多次五折交叉驗證對模型的診斷性能進行評估,多個外部數據集對模型進行驗證。研究流程見圖1。
 圖1
研究的流程圖
圖1
研究的流程圖
ROC:受試者工作特征
1.1 數據下載和處理
從GEO數據庫(https://www.ncbi.nlm.nih.gov/geo/)下載心肌梗死相關數據,以“acute myocardial infarction”為關鍵詞,物種為“homo sapiens”,數據類型為微陣列或高通量測序數據,且數據中應同時包含有對照和心肌梗死數據。共搜索到5個符合條件的相關數據;見表1。下載這些芯片的測序數據及相應的平臺芯片探針信息,在探針ID和基因符號的轉換過程中,若發現多個探針對應于1個基因符號,則以探針表達的平均值用作基因表達水平。鐵死亡基因從FerrDB數據庫(http://www.zhounan.org/ferrdb)獲得,共下載得到259個鐵死亡相關基因。
1.2 篩選AMI中差異表達的鐵死亡基因和功能富集分析
使用“limma”包篩選對照組與AMI中的差異表達基因,按|log2FC|≥1,P≤0.05作為篩選條件。將篩選得到的AMI差異表達基因與鐵死亡相關基因取交集,得到AMI中差異表達的鐵死亡基因。使用Metascape網站(http://metascape.org/)進行基因本體(Gene Ontology,GO)與京都基因與基因組百科全書(Kyoto Encyclopedia of Genes and Genomes,KEGG)功能富集分析,探索鐵死亡基因在調控心肌梗死中潛在的生物學功能。
1.3 隨機森林算法進一步篩選關鍵基因
隨機森林算法是一種監督學習算法,由大量的決策樹構成,對于分類任務具有出色的效果[8]。對得到的差異鐵死亡基因采用隨機森林算法進一步確定具有良好分類性能的關鍵基因。采用R包“randomForest”構建隨機森林模型,計算1~500棵樹的錯誤率和穩定性,以錯誤率最低和穩定性最高的點對應的樹數作為最佳樹數,隨后利用最佳樹數構建隨機森林模型,使用基尼系數得分確定潛在的AMI鐵死亡生物標志物。
1.4 人工神經網絡診斷模型的構建與評估
人工神經網絡是受生物學啟發的算法,旨在模擬人腦神經元處理信息的方式,通常由諸多人工神經元組成,各個神經元之間以計算的權重相連,在醫療領域,人工神經網絡也用于數據的分類、識別、建模和預測等功能[9]。采用外部數據集GSE83500構建人工神經網絡診斷模型,首先對隨機森林算法得到的關鍵基因采用min-max方法進行歸一化,將關鍵基因的表達量轉換為基因評分。將每個樣品中的關鍵基因表達水平與所有樣品的中位值進行比較。如果上調基因的表達水平高于中位值,則將其基因評分定義為1,否則為0。同樣,如果下調基因的表達水平低于中位數,則定義為1,否則為0。采用R包“neuralnet”構建人工神經網絡模型,其包含1個輸入層、1個隱藏層和1個輸出層,各層之間以計算得到的關鍵基因的權重進行連接。使用“caret”包對人工神經網絡模型進行五折交叉驗證[10],將訓練組隨機分為5等份,每次取其中4份進行模型訓練,剩余的1份用于測試,重復上述步驟,最終計算五折交叉曲線下面積(area under the curve,AUC)的平均值評估模型的性能與泛化能力。我們使用“caret”包中的MultiFolds函數進行了10次五折交叉驗證。
1.5 人工神經網絡診斷性能的驗證
使用“pROC”R包繪制了模型組的受試者工作特征(receiver operating characteristic,ROC)曲線并計算AUC值。從GEO數據庫下載了3個外部獨立的數據集驗證特征基因的分類效率,對所有驗證集中的特征基因同樣采用歸一化轉換為基因評分,采用“pROC”包繪制每個驗證集的ROC曲線,計算AUC值驗證分類效率。此外,采用“ggplot”包可視化每個驗證集的混肴矩陣,從另一個方面展示分類效果。
1.6 免疫細胞浸潤分析
單樣本基因集富集分析(single sample gene set enrichment analysis,ssGSEA)能通過將每個樣本的基因表達數據與特定的免疫細胞基因集進行比較,進而來估計每個樣本中的免疫細胞相對豐度[11]。通過R包“GSEABase”“GSVA”和“limma”評估每個樣本的免疫學特征。采用R包“ggplot2”進行免疫細胞之間及免疫細胞與特征基因的相關性分析。
1.7 關鍵基因的藥物預測
以4個關鍵基因為關鍵詞,從醫學本體信息檢索平臺(Coremine Medical)(https://coremine.com/medical/)中篩選出關鍵基因相關的藥物(P<0.05)
1.8 統計學分析
統計學分析采用R軟件(4.2.1)。AMI差異基因的篩選采用t檢驗。檢驗水準α=0.05。
2 結果
2.1 樣本資料
建模組GSE83500數據集共37例樣本,其中男32例、女5例,年齡≤60歲20例,年齡>60歲17例。包括17例AMI和20例非AMI的主動脈血管平滑肌細胞,19014個基因表達信息。
2.2 AMI中鐵死亡差異表達基因的篩選與富集分析
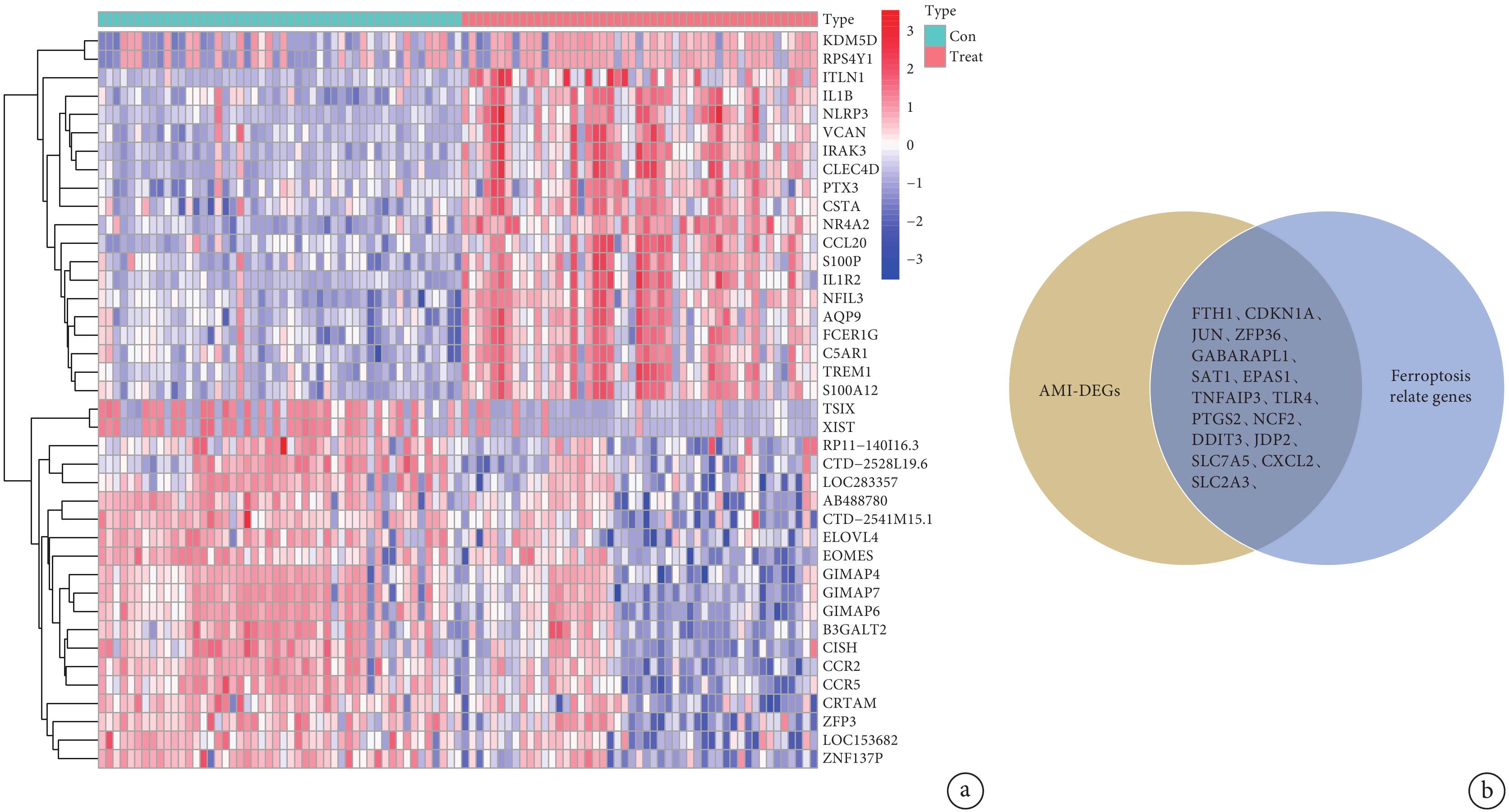
對訓練組進行差異分析共得到365個差異表達基因,其中74個下調,291個上調。從鐵死亡數據庫中共下載得到259個鐵死亡相關基因。取交集后共得到16個AMI中差異表達的鐵死亡基因;見圖2。
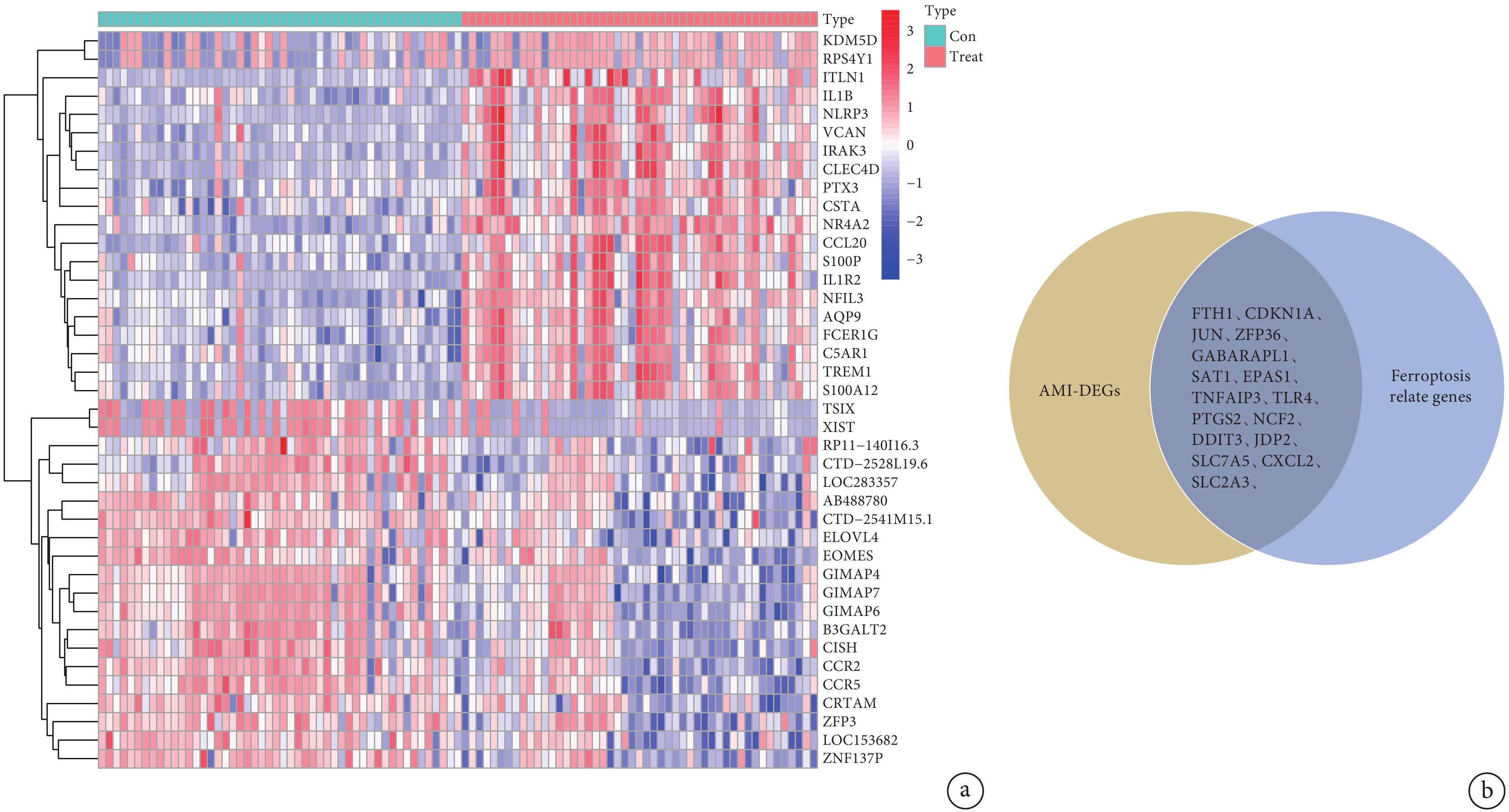 圖2
急性心肌梗死中鐵死亡差異表達基因的篩選
圖2
急性心肌梗死中鐵死亡差異表達基因的篩選
a:訓練組中上調和下調差異最顯著的前20個基因,藍色為低表達,紅色為高表達;b:心肌梗死差異基因與鐵死亡相關基因交集的韋恩圖
2.3 GO與KEGG富集分析
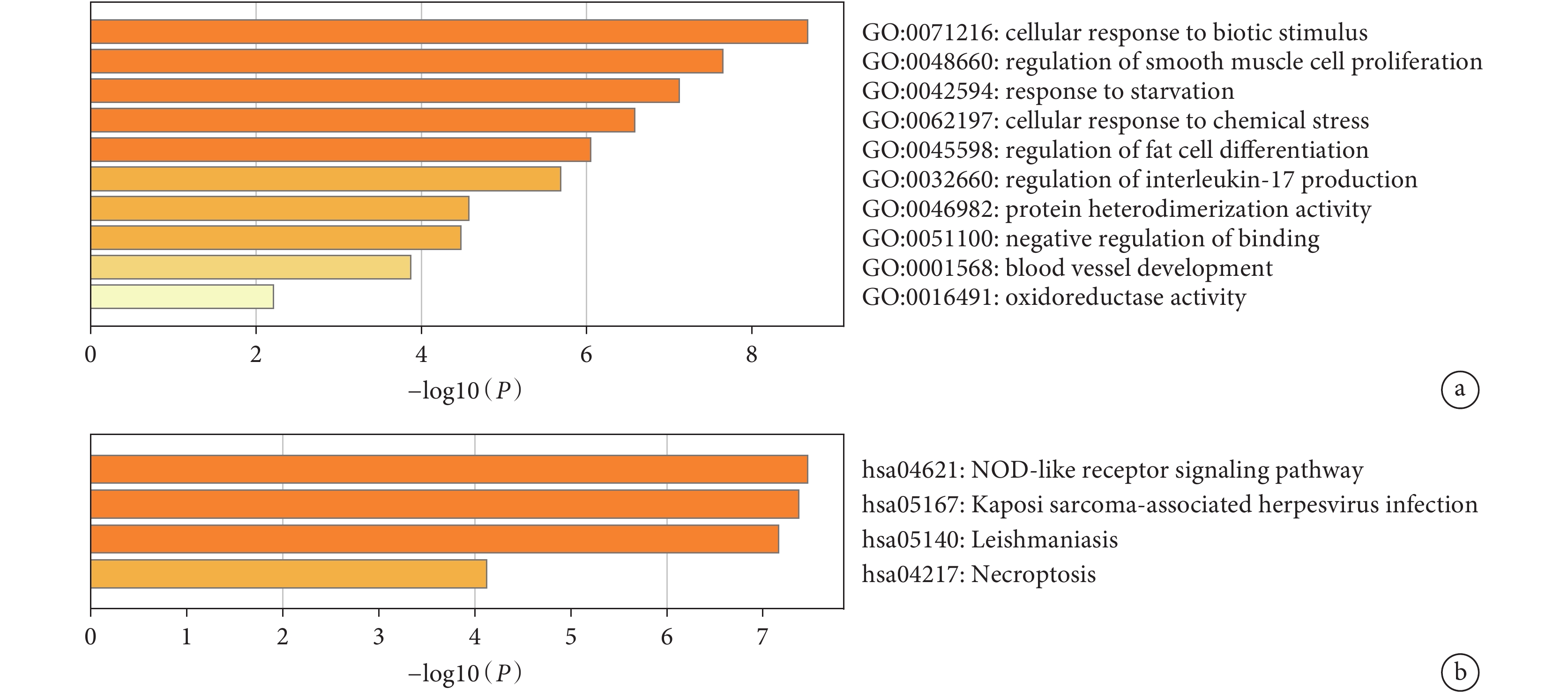
對16個鐵死亡差異表達基因的GO富集分析發現,其主要參與細胞對生物刺激和化學應激的反應、白細胞介素17的調節等生物學功能。同時,這些基因在NOD-like受體信號通路、程序性細胞壞死、利什曼病等途徑顯著富集;見圖3。
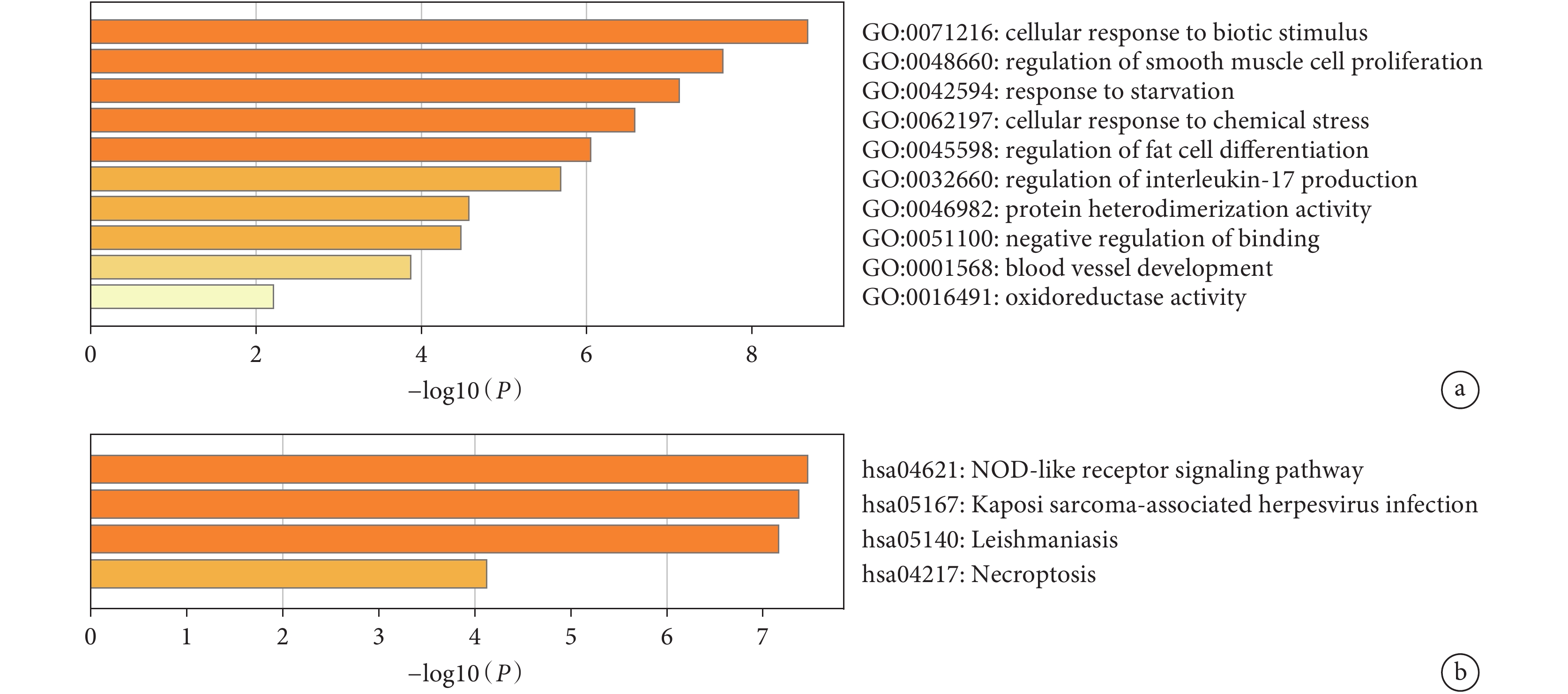 圖3
鐵死亡差異表達基因的富集分析
圖3
鐵死亡差異表達基因的富集分析
a:GO富集分析;b:KEGG富集分析;橫坐標為?log10(
2.4 隨機森林算法鑒定AMI診斷生物標志物
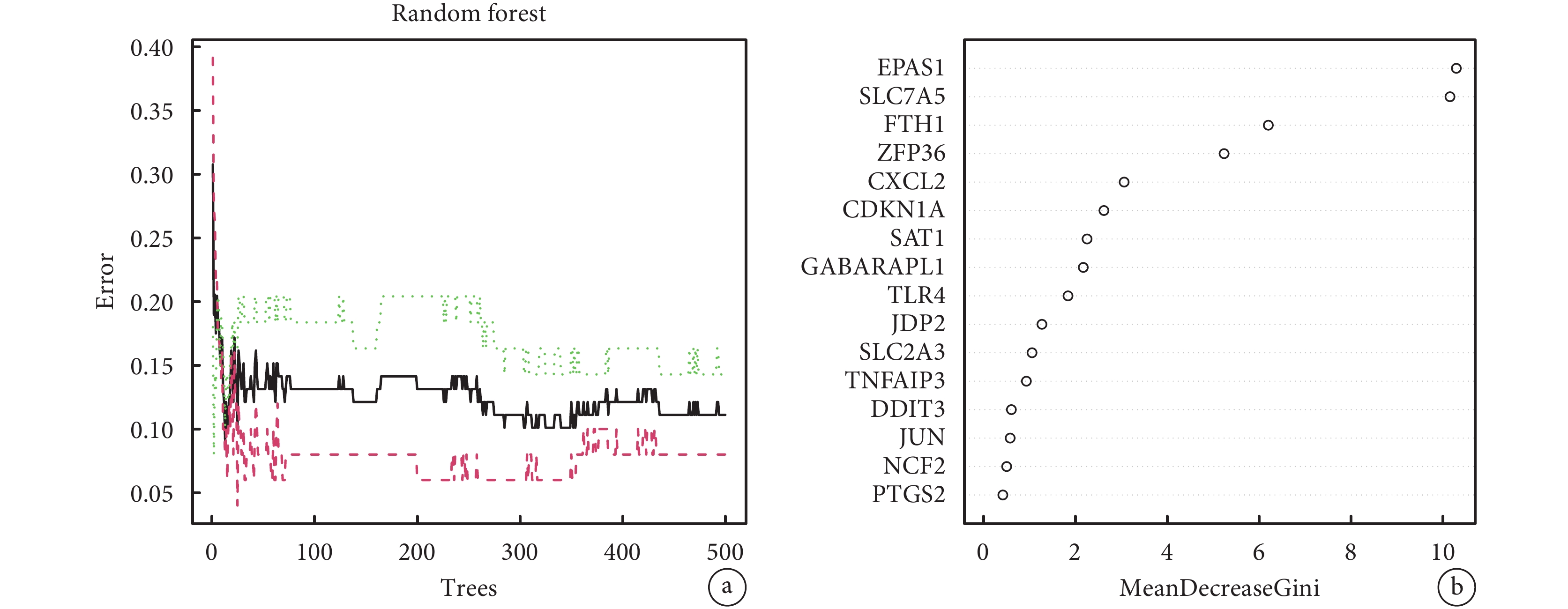
將16個鐵死亡差異表達基因進行隨機森林算法以鑒定具有良好分類性能的特征基因。當trees=13、mtry(節點中二叉樹的最佳變量數)=4時,誤差值相對穩定,此時模型的袋外誤差率最低為11.11%,隨后通過基尼系數法計算每個基因的重要性得分。本研究選擇了基尼系數>4的基因作為后續構建模型的特征基因,分別為EPAS1、SLC7A5、FTH1、ZFP36;見圖4。
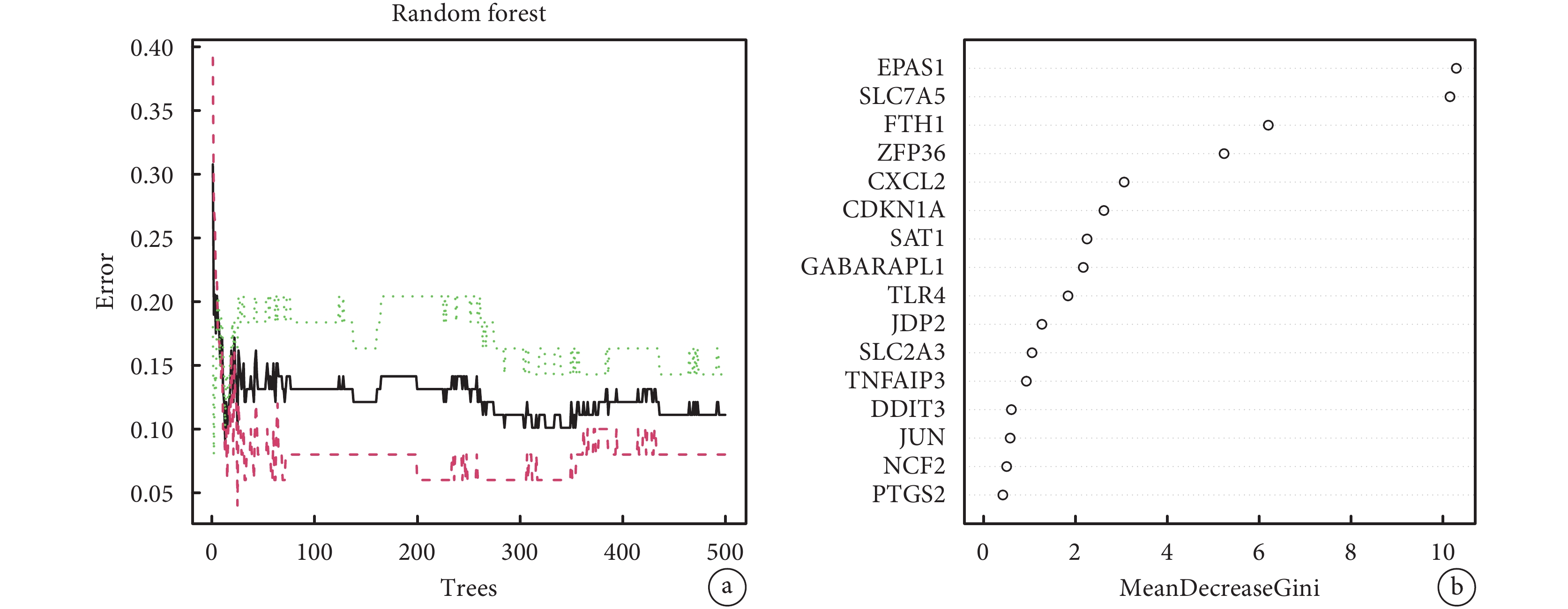 圖4
隨機森林算法篩選特征基因
圖4
隨機森林算法篩選特征基因
a:隨機森林圖,橫坐標代表樹數,縱坐標代表相應樹數對應的袋外誤差率;b:隨機森林算法得到的基尼系數打分
2.5 構建人工神經網絡模型及評估
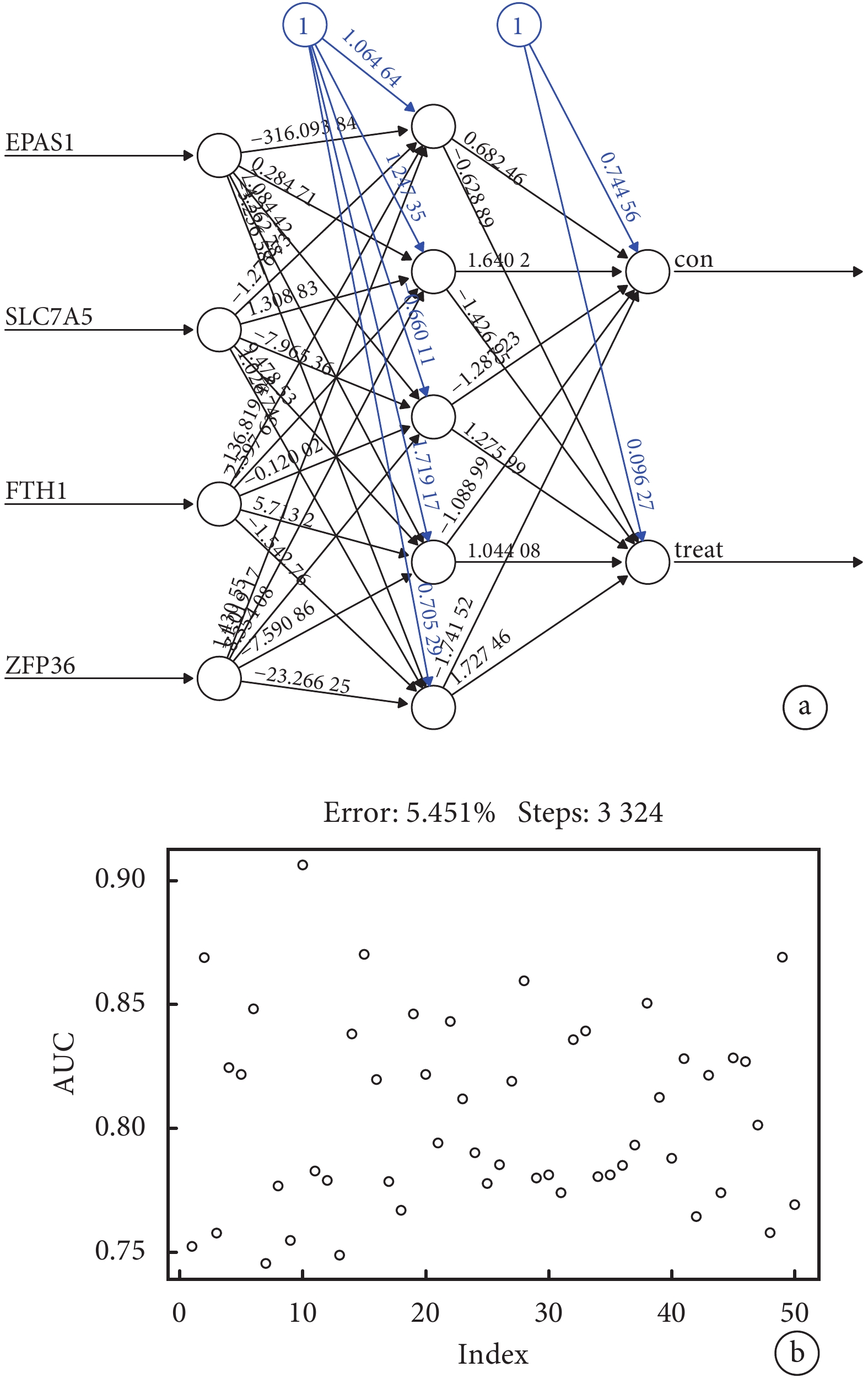
將隨機森林算法得到的4個特征基因作為輸入層構建人工神經網絡模型,4個特征基因已經過min-max方法進行歸一化,隱藏層神經元數默認設為5,輸出層為AMI或正常,各神經元之間由權重相連,權重的計算公式為neuraAMI=Σ(Gene Expression×Neural Network Weight)。結果顯示,神經網絡共運行3324步,錯誤率為5.451%,終止條件為誤差函數的絕對偏導數<0.01。權重值范圍為–316.094~1.431,權重預測值分別為EPAS1(–316.094)、SLC7A5(–1.277)、FTH1(–136.829)、ZFP36(1.431)。對模型進行了10次五折交叉驗證,共計50次交叉驗證,統計50次AUC值發現,其中最小值為0.7456,最大值為0.906,平均值為0.805,這一結果說明此模型具有良好的穩健性;見圖5。
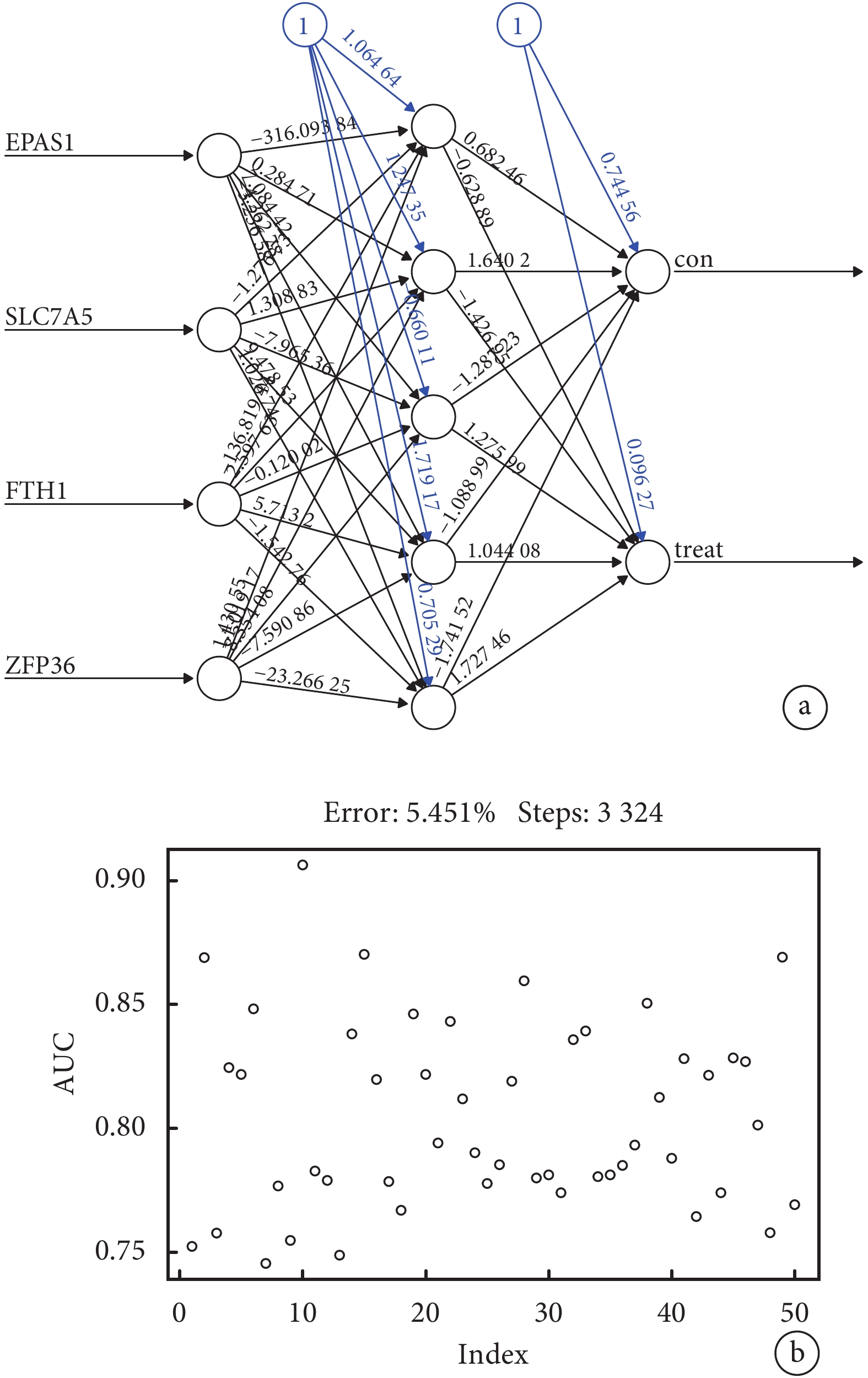 圖5
人工神經網絡模型的構建與評估
圖5
人工神經網絡模型的構建與評估
a:人工神經網絡模型,連線上的數字代表預測的權重;b:10次五折交叉驗證的AUC值散點圖,橫坐標為交叉驗證的次數,縱坐標為對應的AUC值;AUC:曲線下面積
2.6 人工神經網絡模型的驗證
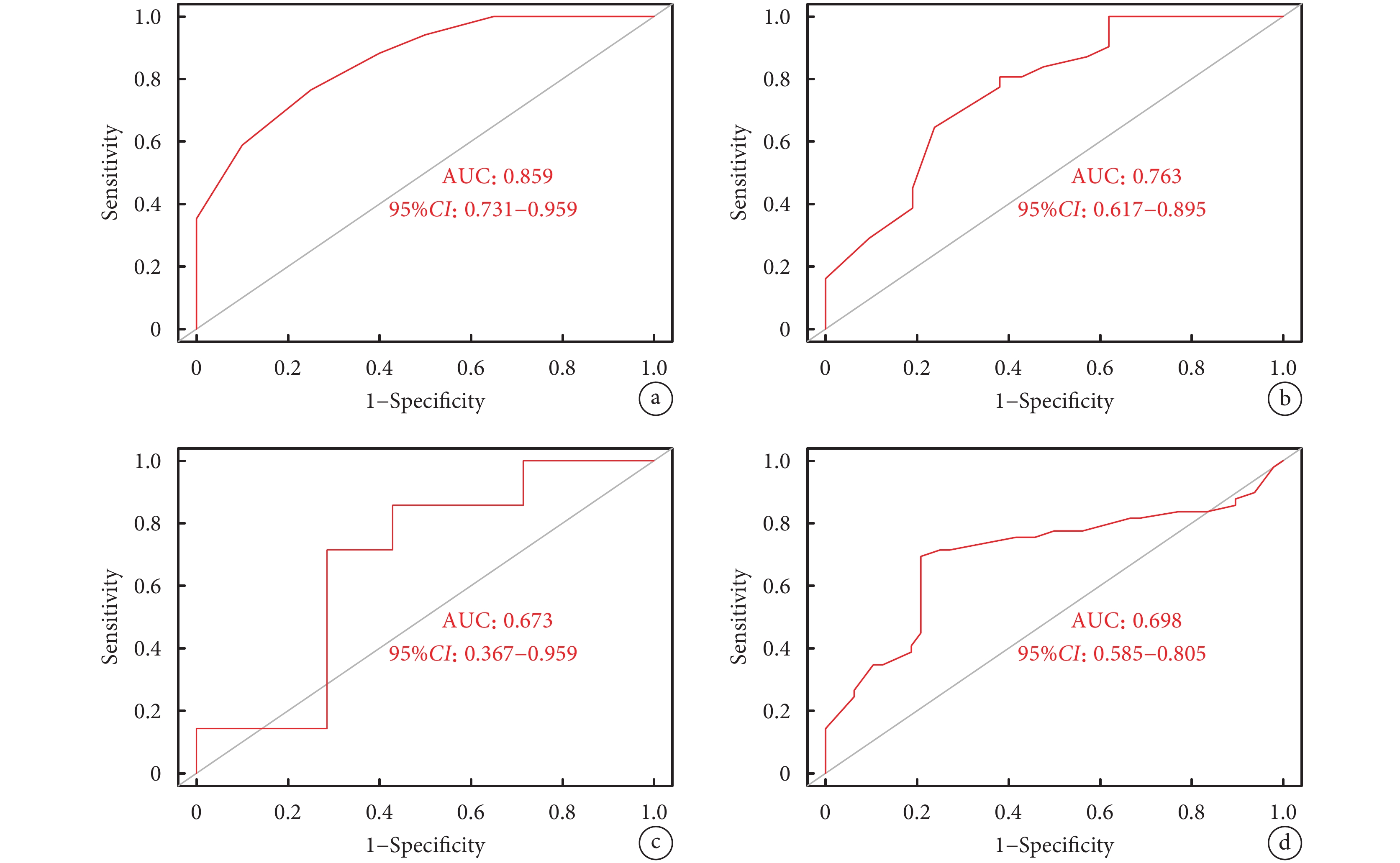
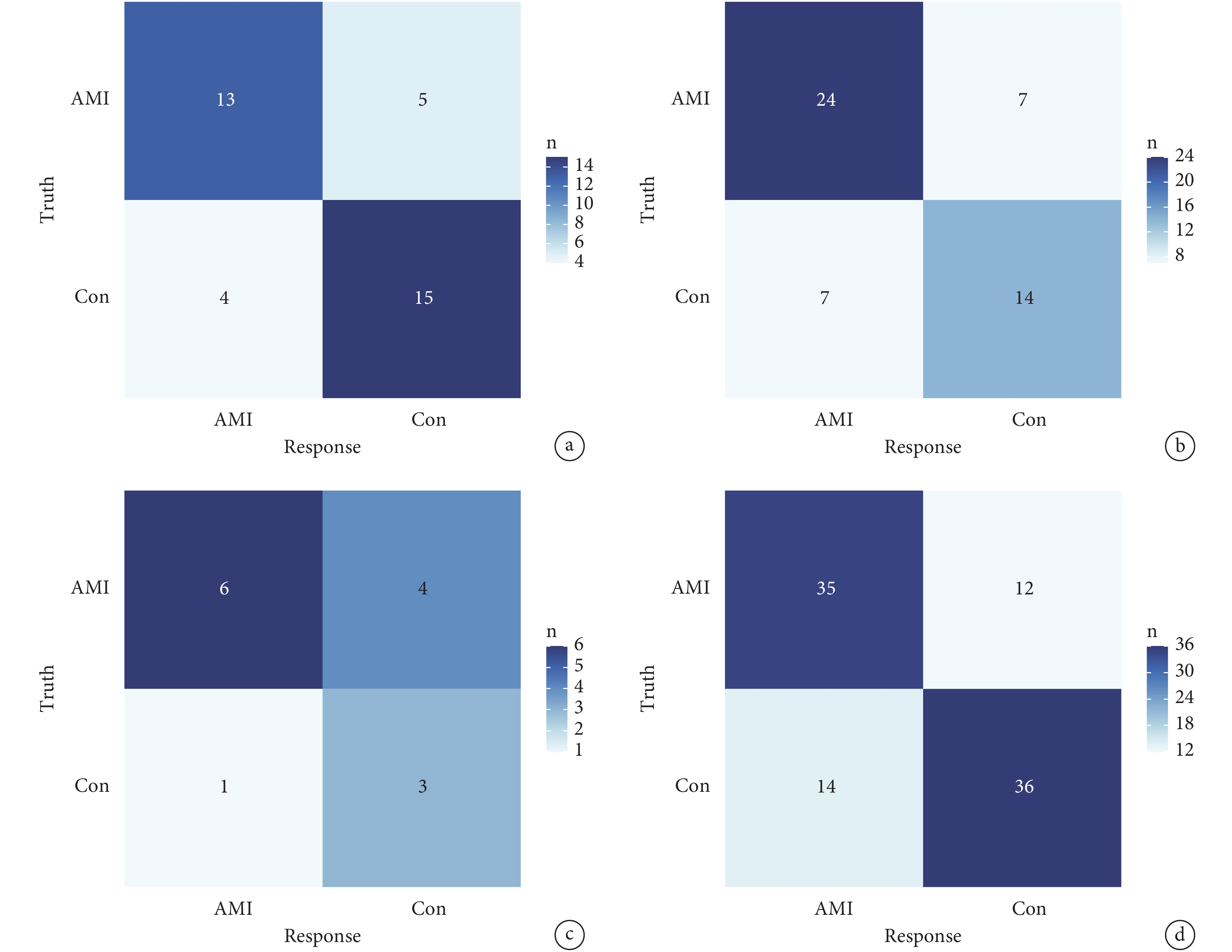
模型組的AUC為0.859,此外,使用3個外部獨立數據集作為驗證組對模型的分類性能進行驗證。結果發現,3個數據集的AUC值分別為0.763(GSE48060)、0.673(GSE60993)、0.698(GSE34198),說明此模型具有一定的診斷性能;見圖6。此外,使用混淆矩陣展示了各驗證組的分類性能;見圖7。
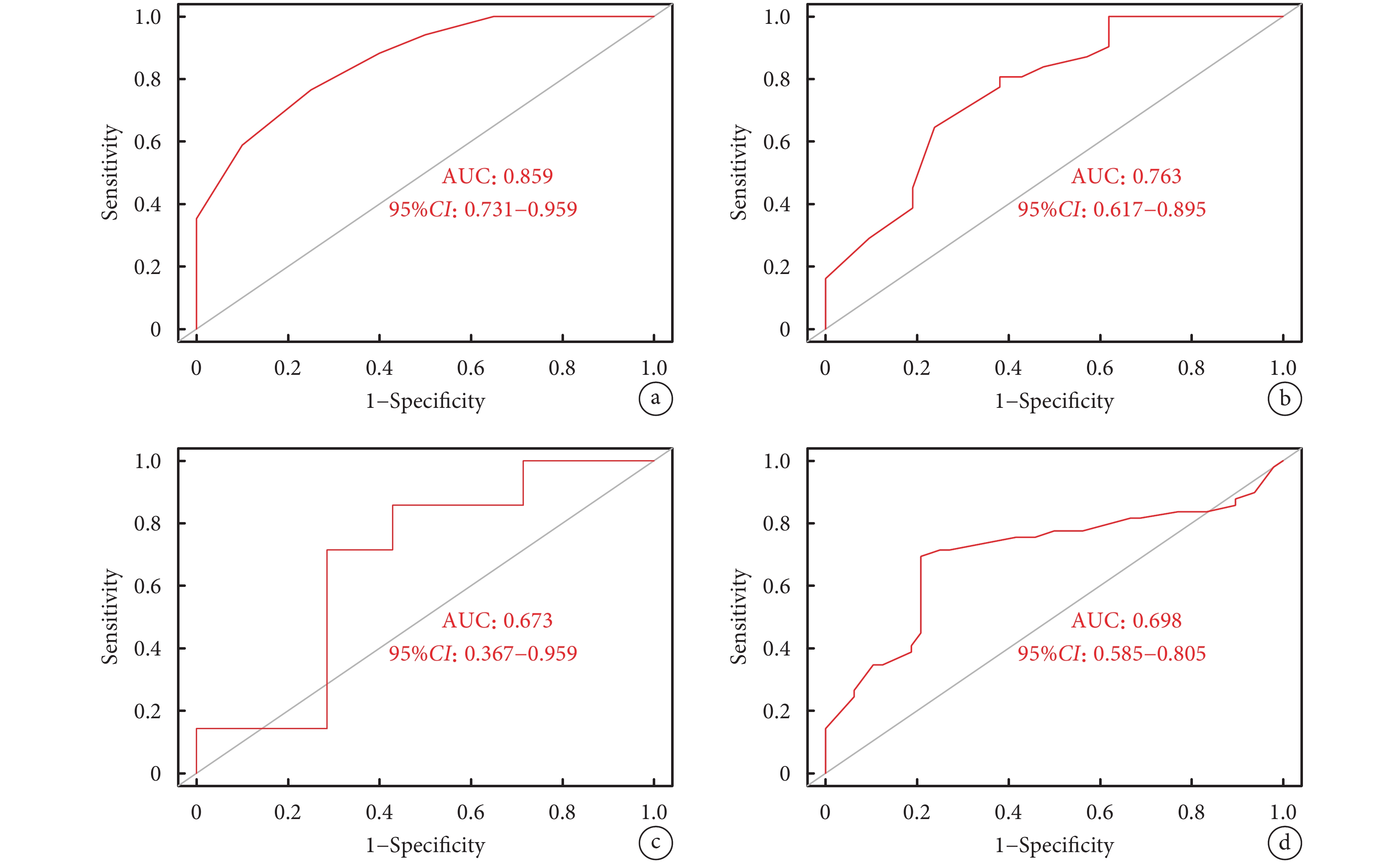 圖6
人工神經網絡模型的驗證
圖6
人工神經網絡模型的驗證
a:模型組的ROC曲線;b~d:驗證組的ROC曲線;ROC:受試者工作特征
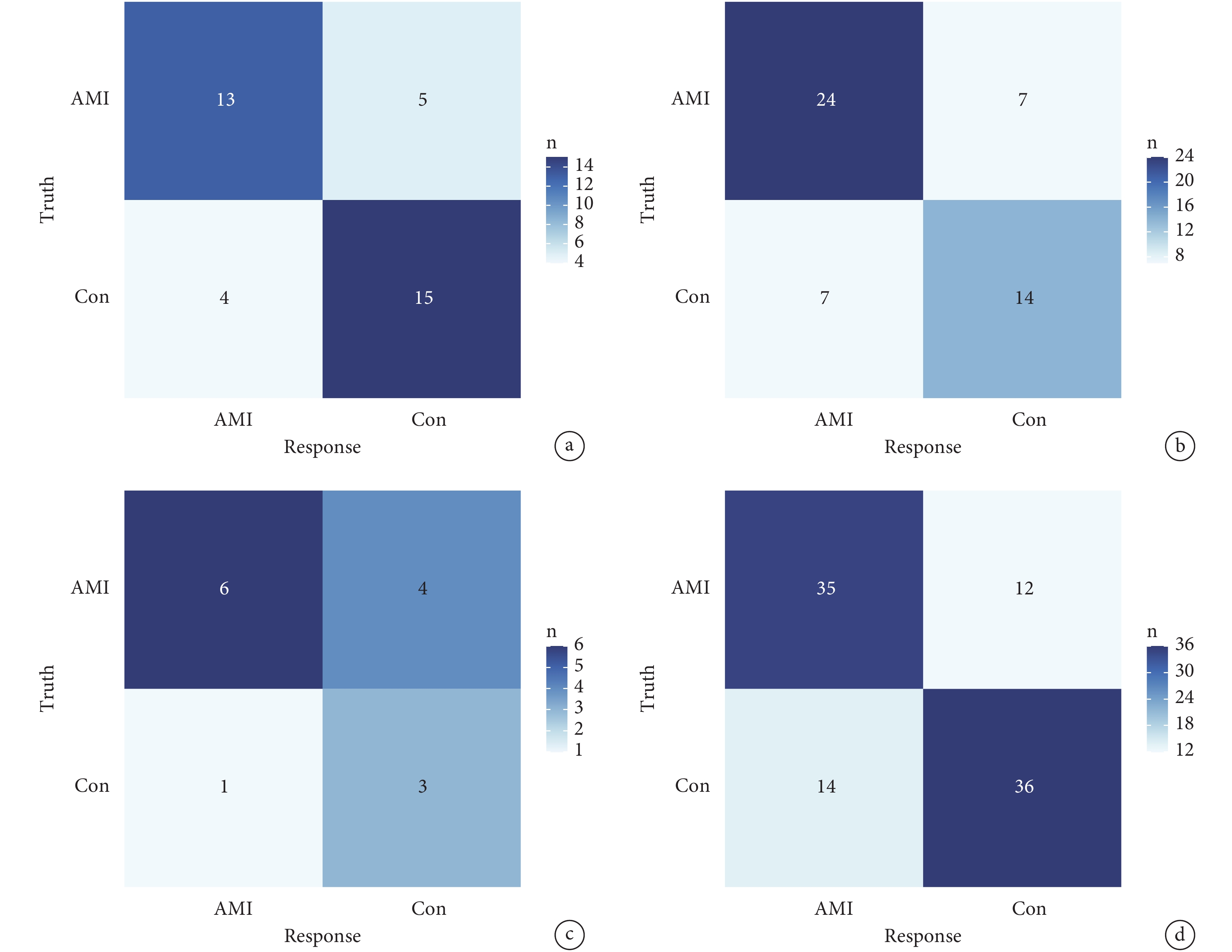 圖7
模型組與驗證組的混淆矩陣
圖7
模型組與驗證組的混淆矩陣
a:模型組的混淆矩陣;b~d:驗證組的混淆矩陣
2.7 免疫細胞浸潤分析
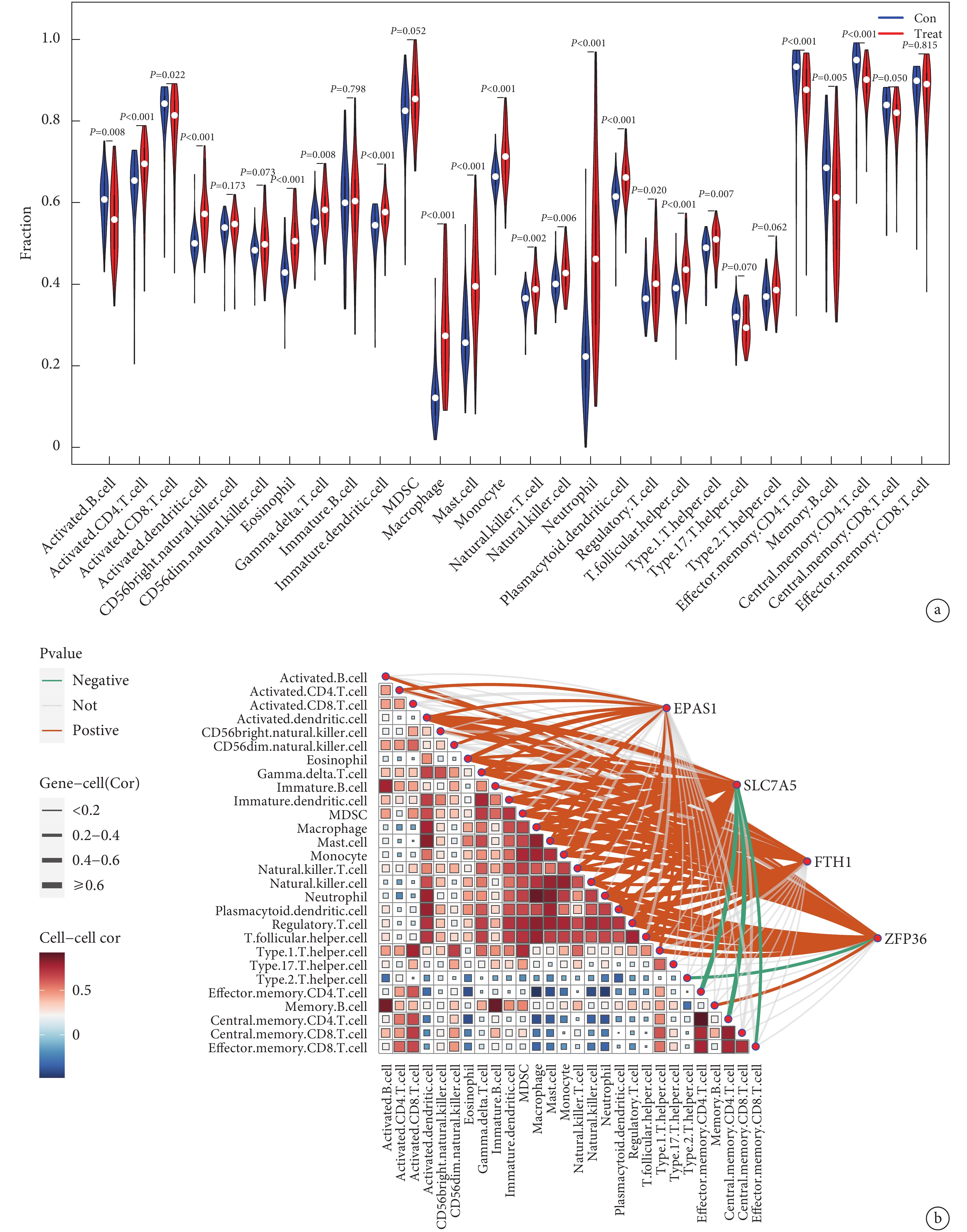
與正常對照組比較,AMI組的巨噬細胞、肥大細胞和單核細胞等顯著活躍,而活化B淋巴細胞、活化CD8+T細胞、中央記憶性CD4+T細胞則受抑制。免疫細胞的相關性分析發現中性粒細胞與巨噬細胞相關性較強,Ⅱ型輔助性T細胞與其他免疫細胞的相關性均較弱。此外,研究還發現4個特征基因與活化樹突狀細胞、嗜酸性粒細胞和γδT細胞等呈正相關;見圖8。
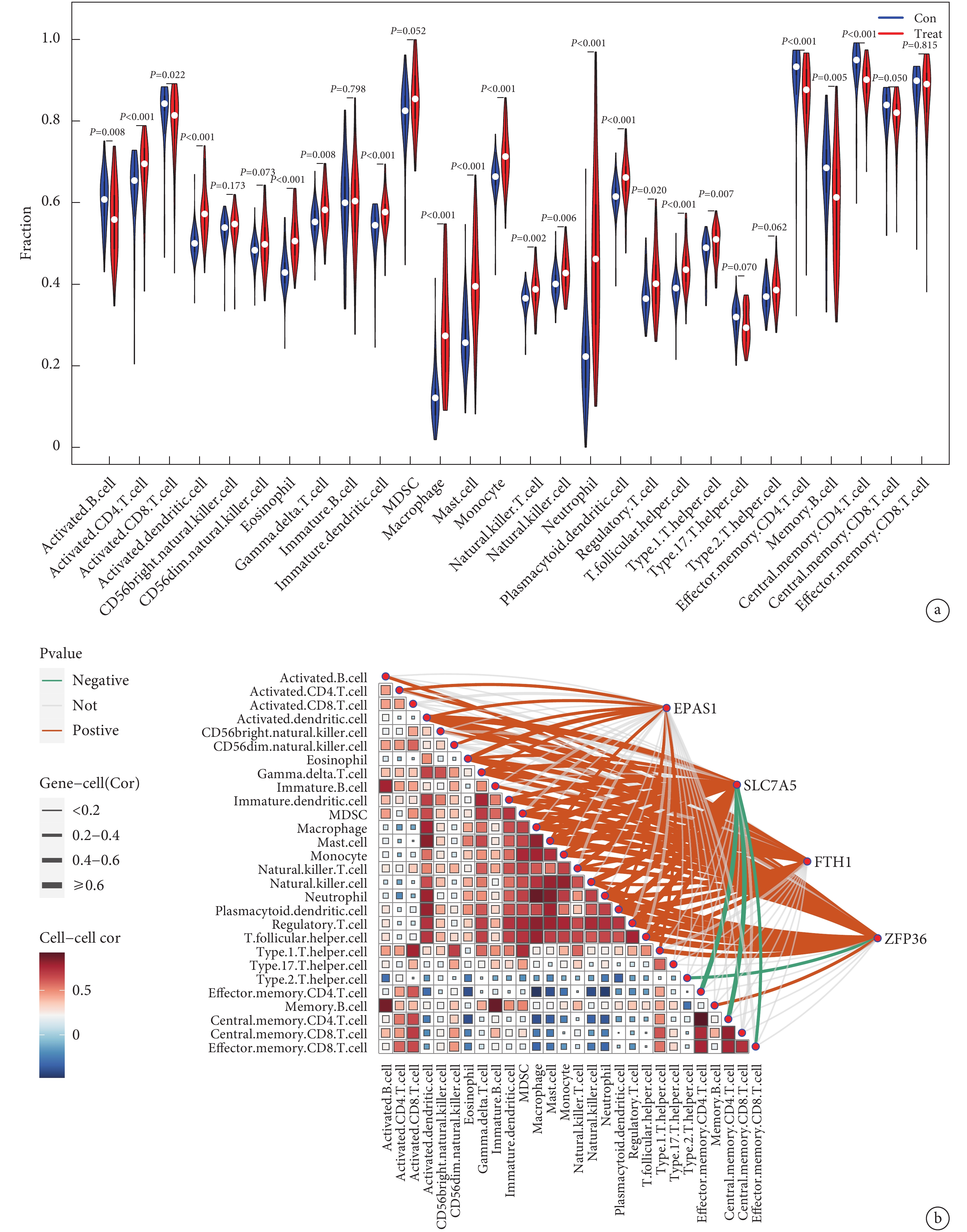 圖8
免疫細胞浸潤分析
圖8
免疫細胞浸潤分析
a:急性心肌梗死中正常與梗死組織的免疫細胞差異情況;b:免疫細胞之間及免疫細胞與特征基因的相關性,橙色連線代表正相關,綠色代表負相關,灰色代表相關性不顯著
2.8 關鍵基因的相關藥物預測
使用Coremine Medical數據庫預測與關鍵基因相關的潛在藥物,共預測到Belzutifan等20種潛在西藥,預測的潛在中藥主要為清熱解毒及活血化瘀類藥物。這些藥物可能通過調節鐵死亡治療AMI;見表2。
3 討論
本研究采用隨機森林結合人工神經網絡構建了AMI鐵死亡相關基因的診斷模型。這種診斷模型也被應用在其他疾病中,包括心力衰竭[12]和子宮內膜異位癥[13]等。鐵死亡參與多種人類疾病的發生與發展,包括心血管疾病相關領域。研究[14]發現,鐵死亡可能與動脈粥樣硬化有關,抑制鐵死亡能夠減弱內皮細胞的脂質過氧化來緩解動脈粥樣硬化。此外,AMI小鼠心肌中miR-26b-5p的下調促進了SLC7A11的表達,從而抑制AMI后鐵死亡并減輕心肌損傷,證明鐵死亡調控中的miR-26b-5p/SLC7A11軸能通過緩解心肌細胞來保護心肌[15]。
基于隨機森林算法篩選出4個用于診斷AMI的鐵死亡基因,分別為EPAS1、SLC7A5、FTH1和ZFP36。內皮PAS結構域包含蛋白-1(EPAS1),也稱為缺氧誘導因子2α(HIF-2α),主要存在于內皮細胞中,研究發現缺氧能夠明顯提高胞內EPAS1蛋白質含量[16]。研究[17]表明,EPAS1在激活血管生成基因的同時也抑制了心肌基因的表達,在成人心肌細胞中敲除EPAS1使心肌細胞重編程成為可能,敲除EPAS1后可以增強成纖維細胞對心肌細胞重編程,從而改善心肌梗死。溶質載體家族7成員5(SLC7A5),也稱為大型中性氨基酸轉運蛋白1(LAT1),是一種跨膜氨基酸轉運蛋白,為細胞和細胞器的生長以及重要的細胞過程提供關鍵氨基酸,從而影響細胞增殖和分化[18]。目前暫無其與AMI相關性的研究,但有研究[19]發現SLC7A5與缺氧密切相關,HIF-2α能夠與SLC7A5的近端啟動子結合促進SLC7A5的表達,這一過程能被多種組織中的缺氧環境所誘導。缺氧可以通過鈣離子超載引起線粒體功能障礙,誘導心肌細胞發生鐵死亡,導致心臟功能受損[20]。因此,在AMI中,我們推測高表達的EPAS1與SLC7A5通過促進鐵死亡過程加重心肌細胞損傷。重肽鐵蛋白1(FTH1)是一種將鐵離子轉運和存儲的蛋白質,負責維持細胞內鐵離子平衡[21]。FTH1能與核受體輔助活化因子4(nuclear receptor coactivator 4,NCOA4)結合通過鐵自噬的方式降解鐵蛋白并釋放大量鐵離子,使胞質內Fe2+進一步升高,從而引起線粒體脂質過氧化和死亡[22]。有文獻[23]報道,環狀RNA(FEACR)能直接與煙酰胺磷酸核糖轉移酶(NAMPT)結合,通過NAMPT-Sirt1-FOXO1-FTH1信號轉導軸上調FTH1來參與心肌細胞鐵死亡的調節,并保護心臟功能免受心肌缺血-再灌注損傷。人鋅指蛋白36(ZFP36)是一種富含AU的元素結合蛋白,近年來被確定為鐵死亡轉錄后調節因子,在肝星狀細胞中,過表達的ZFP36能抑制鐵死亡過程,相反,抑制ZFP36的表達則促進細胞發生鐵死亡[24]。目前暫無ZFP36與AMI的相關研究。我們的研究還發現,ZFP36與諸多免疫細胞呈正相關。有研究[25]發現,在心肌缺血-再灌注后肺部炎癥引起的肺損傷中,敲低ZFP36將導致促炎蛋白(如TNF-α、IL-6)水平升高,從而加重肺損傷。此外,缺乏ZFP36的小鼠也表現出更嚴重的炎癥反應,這與其體內升高的TNF-α有關[26]。結合以上研究,我們推測ZFP36在AMI中高表達,既能抑制心肌細胞發生鐵死亡,也能調集免疫細胞來抑制心肌壞死引起的炎癥。
免疫細胞浸潤分析為我們展示了AMI后的免疫學特征改變。28種免疫細胞差異分析結果顯示AMI后大多數免疫細胞活性增強,其中包括巨噬細胞、單核細胞以及多種淋巴細胞。心肌中的巨噬細胞和單核細胞是心血管疾病中的重要治療靶點,均參與梗死后的免疫應答和炎癥損傷[27]。在AMI的早期階段,心臟損傷會導致單核細胞浸潤該梗死區域,隨后分化為兩種巨噬細胞,其中M1巨噬細胞分泌促炎因子,而M2巨噬細胞分泌抗炎因子[28]。研究[29]發現,在心臟修復期,AMI中最突出的巨噬細胞群是M2樣細胞,其修復蛋白如PDGFB表達較為活躍。淋巴細胞同樣在AMI后的心肌愈合與重塑中發揮重要作用。T淋巴細胞主要為CD4+T細胞,具有多種表型,能激活參與心肌梗死調節的其他免疫細胞發揮免疫作用。研究[30]表明,CD4+T細胞可促進巨噬細胞極化,并有助于減少年輕小鼠的心臟纖維化和增加心肌細胞增殖。
生物信息學的快速發展為AMI的預測提供了新方法。通過生物信息學鑒定了IL1B、CXCL1和CXCL8等生物標志物,可用于預測及診斷AMI[31]。此外,多種生物標志物聯合應用的方法也可顯著提高 AMI 的診斷準確性[32]。我們的研究希望能更進一步完善和補充這些方法,探索更多潛在的AMI診斷生物標志物。隨機森林結合人工神經網絡是一種較為新穎的診斷模型,隨機森林模型可以基于樣本數據構建機器學習模型并用于分類預測,且能評估變量重要性[33]。在人工神經網絡模型中,對關鍵基因進行歸一化評分能去除訓練集和驗證集的批次效應,從而提高模型的預測能力。雖然我們采用了3個獨立數據集進行驗證,但此研究的樣本數量仍較少,可能會影響研究結果的有效性。
鑒于鐵死亡在心血管疾病中有良好的應用前景,我們基于生物信息學篩選了4個具有診斷意義的AMI鐵死亡特征基因,并通過多個公共數據集進行驗證。免疫浸潤分析結果表明AMI后多種免疫細胞表現活躍,參與梗死心肌的愈合及修復。本研究結果進一步為AMI的分子機制研究、生物標志物探索和相關靶向基因藥物提供了有益證據。然而,需要進一步的體外和體內分析實驗來確認與AMI相關的功能通路和樞紐基因。
利益沖突:無。
作者貢獻:鄧海霞負責設計研究;龔玉芳對數據進行分析和撰寫文章;盧燕和周偉負責修改文章。
急性心肌梗死(acute myocardial infarction,AMI)是由冠狀動脈病變引起的臨床病癥,可危及生命,是心臟性猝死的主要原因之一[1]。主要發病原因為冠狀動脈粥樣斑塊破裂,形成血塊阻塞動脈,導致心肌缺血壞死[2]。AMI可分為ST段抬高型心肌梗死和非ST段抬高型心肌梗死[3]。通常,心肌肌鈣蛋白T和心肌肌鈣蛋白I被作為診斷AMI的生物標志物。雖然心肌肌鈣蛋白的增加通常提示心肌壞死,但這也可能是由心力衰竭、心肌炎和胸部創傷等其他疾病引起[4]。所以,我們需要尋找新的診斷AMI的生物標志物,特別是在AMI早期具有高敏感性和特異性的分子,以縮短診斷期并改善AMI患者的預后。
鐵死亡(ferroptosis)是一種不同于細胞凋亡、細胞壞死和自噬的一種細胞死亡方式。主要與細胞內鐵的含量有關,表現為鐵含量過度升高并導致細胞內氧化還原反應失衡、脂質過氧化物的積累和活性氧的產生[5]。隨著冠狀動脈閉塞后血運重建引起再灌注損傷的發展,心肌細胞發生鐵死亡并釋放炎癥介質以加重心臟損傷[6]。有研究[7]發現阿霉素可誘導心臟中的血紅蛋白降解和游離鐵釋放,使心肌細胞發生鐵死亡,從而引發心力衰竭。尋找鐵死亡的新分子靶點正逐漸成為心血管研究領域的焦點。
1 資料與方法
本研究從公共數據庫基因表達綜合數據庫(Gene Expression Omnibus,GEO)下載心肌梗死高通量測序數據,通過隨機森林算法篩選4個具有分類性能的心肌梗死鐵死亡特征基因。通過這4個基因構建人工神經網絡診斷模型。采用多次五折交叉驗證對模型的診斷性能進行評估,多個外部數據集對模型進行驗證。研究流程見圖1。
 圖1
研究的流程圖
圖1
研究的流程圖
ROC:受試者工作特征
1.1 數據下載和處理
從GEO數據庫(https://www.ncbi.nlm.nih.gov/geo/)下載心肌梗死相關數據,以“acute myocardial infarction”為關鍵詞,物種為“homo sapiens”,數據類型為微陣列或高通量測序數據,且數據中應同時包含有對照和心肌梗死數據。共搜索到5個符合條件的相關數據;見表1。下載這些芯片的測序數據及相應的平臺芯片探針信息,在探針ID和基因符號的轉換過程中,若發現多個探針對應于1個基因符號,則以探針表達的平均值用作基因表達水平。鐵死亡基因從FerrDB數據庫(http://www.zhounan.org/ferrdb)獲得,共下載得到259個鐵死亡相關基因。
1.2 篩選AMI中差異表達的鐵死亡基因和功能富集分析
使用“limma”包篩選對照組與AMI中的差異表達基因,按|log2FC|≥1,P≤0.05作為篩選條件。將篩選得到的AMI差異表達基因與鐵死亡相關基因取交集,得到AMI中差異表達的鐵死亡基因。使用Metascape網站(http://metascape.org/)進行基因本體(Gene Ontology,GO)與京都基因與基因組百科全書(Kyoto Encyclopedia of Genes and Genomes,KEGG)功能富集分析,探索鐵死亡基因在調控心肌梗死中潛在的生物學功能。
1.3 隨機森林算法進一步篩選關鍵基因
隨機森林算法是一種監督學習算法,由大量的決策樹構成,對于分類任務具有出色的效果[8]。對得到的差異鐵死亡基因采用隨機森林算法進一步確定具有良好分類性能的關鍵基因。采用R包“randomForest”構建隨機森林模型,計算1~500棵樹的錯誤率和穩定性,以錯誤率最低和穩定性最高的點對應的樹數作為最佳樹數,隨后利用最佳樹數構建隨機森林模型,使用基尼系數得分確定潛在的AMI鐵死亡生物標志物。
1.4 人工神經網絡診斷模型的構建與評估
人工神經網絡是受生物學啟發的算法,旨在模擬人腦神經元處理信息的方式,通常由諸多人工神經元組成,各個神經元之間以計算的權重相連,在醫療領域,人工神經網絡也用于數據的分類、識別、建模和預測等功能[9]。采用外部數據集GSE83500構建人工神經網絡診斷模型,首先對隨機森林算法得到的關鍵基因采用min-max方法進行歸一化,將關鍵基因的表達量轉換為基因評分。將每個樣品中的關鍵基因表達水平與所有樣品的中位值進行比較。如果上調基因的表達水平高于中位值,則將其基因評分定義為1,否則為0。同樣,如果下調基因的表達水平低于中位數,則定義為1,否則為0。采用R包“neuralnet”構建人工神經網絡模型,其包含1個輸入層、1個隱藏層和1個輸出層,各層之間以計算得到的關鍵基因的權重進行連接。使用“caret”包對人工神經網絡模型進行五折交叉驗證[10],將訓練組隨機分為5等份,每次取其中4份進行模型訓練,剩余的1份用于測試,重復上述步驟,最終計算五折交叉曲線下面積(area under the curve,AUC)的平均值評估模型的性能與泛化能力。我們使用“caret”包中的MultiFolds函數進行了10次五折交叉驗證。
1.5 人工神經網絡診斷性能的驗證
使用“pROC”R包繪制了模型組的受試者工作特征(receiver operating characteristic,ROC)曲線并計算AUC值。從GEO數據庫下載了3個外部獨立的數據集驗證特征基因的分類效率,對所有驗證集中的特征基因同樣采用歸一化轉換為基因評分,采用“pROC”包繪制每個驗證集的ROC曲線,計算AUC值驗證分類效率。此外,采用“ggplot”包可視化每個驗證集的混肴矩陣,從另一個方面展示分類效果。
1.6 免疫細胞浸潤分析
單樣本基因集富集分析(single sample gene set enrichment analysis,ssGSEA)能通過將每個樣本的基因表達數據與特定的免疫細胞基因集進行比較,進而來估計每個樣本中的免疫細胞相對豐度[11]。通過R包“GSEABase”“GSVA”和“limma”評估每個樣本的免疫學特征。采用R包“ggplot2”進行免疫細胞之間及免疫細胞與特征基因的相關性分析。
1.7 關鍵基因的藥物預測
以4個關鍵基因為關鍵詞,從醫學本體信息檢索平臺(Coremine Medical)(https://coremine.com/medical/)中篩選出關鍵基因相關的藥物(P<0.05)
1.8 統計學分析
統計學分析采用R軟件(4.2.1)。AMI差異基因的篩選采用t檢驗。檢驗水準α=0.05。
2 結果
2.1 樣本資料
建模組GSE83500數據集共37例樣本,其中男32例、女5例,年齡≤60歲20例,年齡>60歲17例。包括17例AMI和20例非AMI的主動脈血管平滑肌細胞,19014個基因表達信息。
2.2 AMI中鐵死亡差異表達基因的篩選與富集分析
對訓練組進行差異分析共得到365個差異表達基因,其中74個下調,291個上調。從鐵死亡數據庫中共下載得到259個鐵死亡相關基因。取交集后共得到16個AMI中差異表達的鐵死亡基因;見圖2。
 圖2
急性心肌梗死中鐵死亡差異表達基因的篩選
圖2
急性心肌梗死中鐵死亡差異表達基因的篩選
a:訓練組中上調和下調差異最顯著的前20個基因,藍色為低表達,紅色為高表達;b:心肌梗死差異基因與鐵死亡相關基因交集的韋恩圖
2.3 GO與KEGG富集分析
對16個鐵死亡差異表達基因的GO富集分析發現,其主要參與細胞對生物刺激和化學應激的反應、白細胞介素17的調節等生物學功能。同時,這些基因在NOD-like受體信號通路、程序性細胞壞死、利什曼病等途徑顯著富集;見圖3。
 圖3
鐵死亡差異表達基因的富集分析
圖3
鐵死亡差異表達基因的富集分析
a:GO富集分析;b:KEGG富集分析;橫坐標為?log10(
2.4 隨機森林算法鑒定AMI診斷生物標志物
將16個鐵死亡差異表達基因進行隨機森林算法以鑒定具有良好分類性能的特征基因。當trees=13、mtry(節點中二叉樹的最佳變量數)=4時,誤差值相對穩定,此時模型的袋外誤差率最低為11.11%,隨后通過基尼系數法計算每個基因的重要性得分。本研究選擇了基尼系數>4的基因作為后續構建模型的特征基因,分別為EPAS1、SLC7A5、FTH1、ZFP36;見圖4。
 圖4
隨機森林算法篩選特征基因
圖4
隨機森林算法篩選特征基因
a:隨機森林圖,橫坐標代表樹數,縱坐標代表相應樹數對應的袋外誤差率;b:隨機森林算法得到的基尼系數打分
2.5 構建人工神經網絡模型及評估
將隨機森林算法得到的4個特征基因作為輸入層構建人工神經網絡模型,4個特征基因已經過min-max方法進行歸一化,隱藏層神經元數默認設為5,輸出層為AMI或正常,各神經元之間由權重相連,權重的計算公式為neuraAMI=Σ(Gene Expression×Neural Network Weight)。結果顯示,神經網絡共運行3324步,錯誤率為5.451%,終止條件為誤差函數的絕對偏導數<0.01。權重值范圍為–316.094~1.431,權重預測值分別為EPAS1(–316.094)、SLC7A5(–1.277)、FTH1(–136.829)、ZFP36(1.431)。對模型進行了10次五折交叉驗證,共計50次交叉驗證,統計50次AUC值發現,其中最小值為0.7456,最大值為0.906,平均值為0.805,這一結果說明此模型具有良好的穩健性;見圖5。
 圖5
人工神經網絡模型的構建與評估
圖5
人工神經網絡模型的構建與評估
a:人工神經網絡模型,連線上的數字代表預測的權重;b:10次五折交叉驗證的AUC值散點圖,橫坐標為交叉驗證的次數,縱坐標為對應的AUC值;AUC:曲線下面積
2.6 人工神經網絡模型的驗證
模型組的AUC為0.859,此外,使用3個外部獨立數據集作為驗證組對模型的分類性能進行驗證。結果發現,3個數據集的AUC值分別為0.763(GSE48060)、0.673(GSE60993)、0.698(GSE34198),說明此模型具有一定的診斷性能;見圖6。此外,使用混淆矩陣展示了各驗證組的分類性能;見圖7。
 圖6
人工神經網絡模型的驗證
圖6
人工神經網絡模型的驗證
a:模型組的ROC曲線;b~d:驗證組的ROC曲線;ROC:受試者工作特征
 圖7
模型組與驗證組的混淆矩陣
圖7
模型組與驗證組的混淆矩陣
a:模型組的混淆矩陣;b~d:驗證組的混淆矩陣
2.7 免疫細胞浸潤分析
與正常對照組比較,AMI組的巨噬細胞、肥大細胞和單核細胞等顯著活躍,而活化B淋巴細胞、活化CD8+T細胞、中央記憶性CD4+T細胞則受抑制。免疫細胞的相關性分析發現中性粒細胞與巨噬細胞相關性較強,Ⅱ型輔助性T細胞與其他免疫細胞的相關性均較弱。此外,研究還發現4個特征基因與活化樹突狀細胞、嗜酸性粒細胞和γδT細胞等呈正相關;見圖8。
 圖8
免疫細胞浸潤分析
圖8
免疫細胞浸潤分析
a:急性心肌梗死中正常與梗死組織的免疫細胞差異情況;b:免疫細胞之間及免疫細胞與特征基因的相關性,橙色連線代表正相關,綠色代表負相關,灰色代表相關性不顯著
2.8 關鍵基因的相關藥物預測
使用Coremine Medical數據庫預測與關鍵基因相關的潛在藥物,共預測到Belzutifan等20種潛在西藥,預測的潛在中藥主要為清熱解毒及活血化瘀類藥物。這些藥物可能通過調節鐵死亡治療AMI;見表2。
3 討論
本研究采用隨機森林結合人工神經網絡構建了AMI鐵死亡相關基因的診斷模型。這種診斷模型也被應用在其他疾病中,包括心力衰竭[12]和子宮內膜異位癥[13]等。鐵死亡參與多種人類疾病的發生與發展,包括心血管疾病相關領域。研究[14]發現,鐵死亡可能與動脈粥樣硬化有關,抑制鐵死亡能夠減弱內皮細胞的脂質過氧化來緩解動脈粥樣硬化。此外,AMI小鼠心肌中miR-26b-5p的下調促進了SLC7A11的表達,從而抑制AMI后鐵死亡并減輕心肌損傷,證明鐵死亡調控中的miR-26b-5p/SLC7A11軸能通過緩解心肌細胞來保護心肌[15]。
基于隨機森林算法篩選出4個用于診斷AMI的鐵死亡基因,分別為EPAS1、SLC7A5、FTH1和ZFP36。內皮PAS結構域包含蛋白-1(EPAS1),也稱為缺氧誘導因子2α(HIF-2α),主要存在于內皮細胞中,研究發現缺氧能夠明顯提高胞內EPAS1蛋白質含量[16]。研究[17]表明,EPAS1在激活血管生成基因的同時也抑制了心肌基因的表達,在成人心肌細胞中敲除EPAS1使心肌細胞重編程成為可能,敲除EPAS1后可以增強成纖維細胞對心肌細胞重編程,從而改善心肌梗死。溶質載體家族7成員5(SLC7A5),也稱為大型中性氨基酸轉運蛋白1(LAT1),是一種跨膜氨基酸轉運蛋白,為細胞和細胞器的生長以及重要的細胞過程提供關鍵氨基酸,從而影響細胞增殖和分化[18]。目前暫無其與AMI相關性的研究,但有研究[19]發現SLC7A5與缺氧密切相關,HIF-2α能夠與SLC7A5的近端啟動子結合促進SLC7A5的表達,這一過程能被多種組織中的缺氧環境所誘導。缺氧可以通過鈣離子超載引起線粒體功能障礙,誘導心肌細胞發生鐵死亡,導致心臟功能受損[20]。因此,在AMI中,我們推測高表達的EPAS1與SLC7A5通過促進鐵死亡過程加重心肌細胞損傷。重肽鐵蛋白1(FTH1)是一種將鐵離子轉運和存儲的蛋白質,負責維持細胞內鐵離子平衡[21]。FTH1能與核受體輔助活化因子4(nuclear receptor coactivator 4,NCOA4)結合通過鐵自噬的方式降解鐵蛋白并釋放大量鐵離子,使胞質內Fe2+進一步升高,從而引起線粒體脂質過氧化和死亡[22]。有文獻[23]報道,環狀RNA(FEACR)能直接與煙酰胺磷酸核糖轉移酶(NAMPT)結合,通過NAMPT-Sirt1-FOXO1-FTH1信號轉導軸上調FTH1來參與心肌細胞鐵死亡的調節,并保護心臟功能免受心肌缺血-再灌注損傷。人鋅指蛋白36(ZFP36)是一種富含AU的元素結合蛋白,近年來被確定為鐵死亡轉錄后調節因子,在肝星狀細胞中,過表達的ZFP36能抑制鐵死亡過程,相反,抑制ZFP36的表達則促進細胞發生鐵死亡[24]。目前暫無ZFP36與AMI的相關研究。我們的研究還發現,ZFP36與諸多免疫細胞呈正相關。有研究[25]發現,在心肌缺血-再灌注后肺部炎癥引起的肺損傷中,敲低ZFP36將導致促炎蛋白(如TNF-α、IL-6)水平升高,從而加重肺損傷。此外,缺乏ZFP36的小鼠也表現出更嚴重的炎癥反應,這與其體內升高的TNF-α有關[26]。結合以上研究,我們推測ZFP36在AMI中高表達,既能抑制心肌細胞發生鐵死亡,也能調集免疫細胞來抑制心肌壞死引起的炎癥。
免疫細胞浸潤分析為我們展示了AMI后的免疫學特征改變。28種免疫細胞差異分析結果顯示AMI后大多數免疫細胞活性增強,其中包括巨噬細胞、單核細胞以及多種淋巴細胞。心肌中的巨噬細胞和單核細胞是心血管疾病中的重要治療靶點,均參與梗死后的免疫應答和炎癥損傷[27]。在AMI的早期階段,心臟損傷會導致單核細胞浸潤該梗死區域,隨后分化為兩種巨噬細胞,其中M1巨噬細胞分泌促炎因子,而M2巨噬細胞分泌抗炎因子[28]。研究[29]發現,在心臟修復期,AMI中最突出的巨噬細胞群是M2樣細胞,其修復蛋白如PDGFB表達較為活躍。淋巴細胞同樣在AMI后的心肌愈合與重塑中發揮重要作用。T淋巴細胞主要為CD4+T細胞,具有多種表型,能激活參與心肌梗死調節的其他免疫細胞發揮免疫作用。研究[30]表明,CD4+T細胞可促進巨噬細胞極化,并有助于減少年輕小鼠的心臟纖維化和增加心肌細胞增殖。
生物信息學的快速發展為AMI的預測提供了新方法。通過生物信息學鑒定了IL1B、CXCL1和CXCL8等生物標志物,可用于預測及診斷AMI[31]。此外,多種生物標志物聯合應用的方法也可顯著提高 AMI 的診斷準確性[32]。我們的研究希望能更進一步完善和補充這些方法,探索更多潛在的AMI診斷生物標志物。隨機森林結合人工神經網絡是一種較為新穎的診斷模型,隨機森林模型可以基于樣本數據構建機器學習模型并用于分類預測,且能評估變量重要性[33]。在人工神經網絡模型中,對關鍵基因進行歸一化評分能去除訓練集和驗證集的批次效應,從而提高模型的預測能力。雖然我們采用了3個獨立數據集進行驗證,但此研究的樣本數量仍較少,可能會影響研究結果的有效性。
鑒于鐵死亡在心血管疾病中有良好的應用前景,我們基于生物信息學篩選了4個具有診斷意義的AMI鐵死亡特征基因,并通過多個公共數據集進行驗證。免疫浸潤分析結果表明AMI后多種免疫細胞表現活躍,參與梗死心肌的愈合及修復。本研究結果進一步為AMI的分子機制研究、生物標志物探索和相關靶向基因藥物提供了有益證據。然而,需要進一步的體外和體內分析實驗來確認與AMI相關的功能通路和樞紐基因。
利益沖突:無。
作者貢獻:鄧海霞負責設計研究;龔玉芳對數據進行分析和撰寫文章;盧燕和周偉負責修改文章。